
Fylogenytolkning, treslag, applikasjoner
EN fylogeni, I evolusjonsbiologi er det en representasjon av evolusjonshistorien til en gruppe organismer eller en art, med vekt på nedstigningslinjen og slektsforholdet mellom gruppene..
I dag har biologer brukt data primært fra komparativ morfologi og anatomi, og fra gensekvenser for å rekonstruere tusenvis av tusenvis av trær..
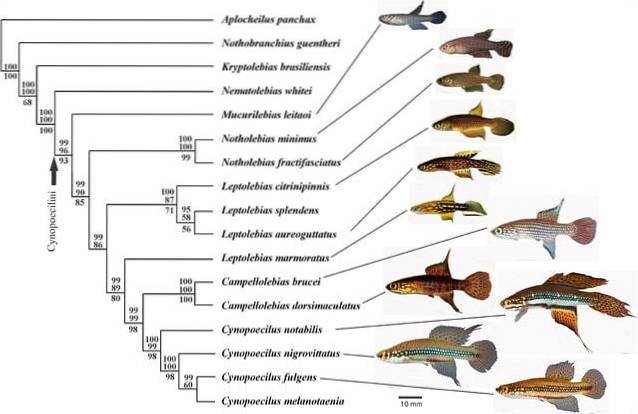
Disse trærne søker å beskrive evolusjonshistorien til de forskjellige dyreartene, planter, mikrober og andre organiske vesener som bor på jorden..
Analogien med livets tre stammer fra Charles Darwins tid. Denne strålende britiske naturforskeren fanger opp mesterverket "Opprinnelsen til arter"Et enkelt bilde: et" tre "som representerer forgreningen av linjene, med utgangspunkt i en felles forfader.
Artikkelindeks
- 1 Hva er fylogeni?
- 2 Hva er et fylogenetisk tre?
- 3 Hvordan tolkes fylogenetiske trær?
- 4 Hvordan rekonstrueres fylogenier?
- 4.1 Homologe tegn
- 5 Typer trær
- 6 Politomias
- 7 Evolusjonær klassifisering
- 7.1 Monofyletiske linjer
- 7.2 Parafyletiske og polyfyletiske linjer
- 8 Søknader
- 9 Referanser
Hva er fylogeni?
I lys av de biologiske vitenskapene er evolusjon en av de mest fantastiske hendelsene som har funnet sted. Denne endringen i organiske former over tid kan representeres i et fylogenetisk tre. Derfor uttrykker fylogeni historien til slekter og hvordan de har endret seg over tid..
En av de direkte implikasjonene av denne grafen er vanlig herkomst. Det vil si at alle organismer som vi ser i dag har dukket opp som etterkommere med modifikasjoner av tidligere former. Denne ideen har vært en av de viktigste i vitenskapshistorien.
Alle livsformene vi kan sette pris på i dag - fra mikroskopiske bakterier, til planter og de største virveldyrene - henger sammen, og dette forholdet er representert i det store og intrikate livets tre..
I samsvar med treet, ville arten som lever i dag representere bladene og resten av grenene være deres evolusjonære historie.
Hva er et fylogenetisk tre?
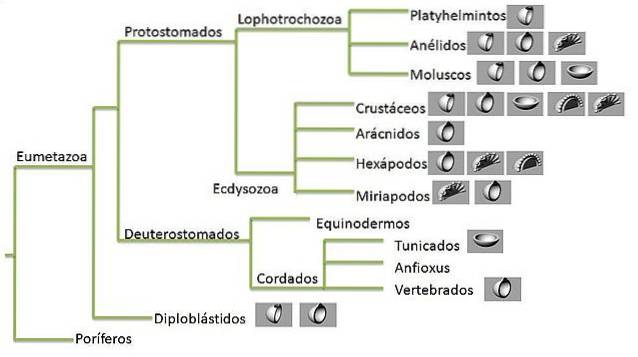
Et fylogenetisk tre er en grafisk fremstilling av evolusjonshistorien til en gruppe organismer. Dette mønsteret av historiske forhold er fylogenien som forskere prøver å estimere..
Trær består av noder som forbinder "grenene". Terminalknutepunktene til hver gren er terminal taxa og representerer sekvensene eller organismer som data er kjent for - disse kan være levende eller utdøde arter.
De interne nodene representerer hypotetiske forfedre, mens stamfaren som er funnet ved roten av treet representerer forfedren til alle sekvensene som er representert i grafen..
Hvordan tolkes fylogenetiske trær?
Det er mange måter å representere et fylogenetisk tre på. Derfor er det viktig å vite hvordan man skal gjenkjenne om disse forskjellene som observeres mellom to trær, skyldes forskjellige topologier - det vil si reelle forskjeller som tilsvarer to stavemåter - eller rett og slett er forskjeller relatert til representasjonsstilen..
For eksempel kan rekkefølgen etikettene vises øverst variere, uten å endre betydningen av den grafiske representasjonen, generelt navnet på arten, slekten, familien, blant andre kategorier..
Dette skjer fordi trærne ligner en mobil, der grenene kan rotere uten å endre forholdet til den representerte arten..
Slik sett spiller det ingen rolle hvor mange ganger rekkefølgen endres eller gjenstandene som "henger" roteres, siden det ikke endrer måten de kobles på - og det er det viktige..
Hvordan rekonstrueres fylogenier?
Fylogenier er hypoteser som er formulert basert på indirekte bevis. Å belyse en fylogeni ligner jobben til en etterforsker for å løse en forbrytelse ved å følge ledetrådene fra åstedet.
Biologer postulerer ofte fylogeniene sine ved hjelp av kunnskap fra forskjellige grener, som paleontologi, komparativ anatomi, komparativ embryologi og molekylærbiologi..
Fossilregistreringen, selv om den er ufullstendig, gir svært verdifull informasjon om avvikstidene for grupper av arter.
Med tiden har molekylærbiologi overgått alle de ovennevnte feltene, og de fleste fylogenier er utledet fra molekylære data..
Målet med å rekonstruere et fylogenetisk tre har en rekke store ulemper. Det er omtrent 1,8 millioner navngitte arter og mange flere uten å bli beskrevet.
Og selv om et betydelig antall forskere prøver å rekonstruere forholdet mellom arter hver dag, er det fortsatt ikke noe komplett tre.
Homologe karakterer
Når biologer ønsker å beskrive likhetene mellom to strukturer eller prosesser, kan de gjøre det når det gjelder felles forfedre (homologier), analogier (funksjon) eller homoplasi (morfologisk likhet)..
For å rekonstruere en fylogeni, brukes utelukkende homologe tegn. Homologi er et nøkkelbegrep i evolusjon og i rekreasjon av forhold mellom arter, siden det bare gjenspeiler tilstrekkelig organismenes felles forfedre..
Anta at vi ønsker å utlede fylogenien til tre grupper: fugler, flaggermus og mennesker. For å oppfylle vårt mål bestemte vi oss for å bruke øvre ekstremiteter som et kjennetegn som hjelper oss å skille mønsteret av forhold..
Siden fugler og flaggermus har modifisert strukturer for flukt, kan vi feilaktig konkludere med at flaggermus og fugler er nærmere knyttet til hverandre enn flaggermus til mennesker. Hvorfor har vi kommet til feil konklusjon? Fordi vi har brukt en analog og ikke-homolog karakter.
For å finne det rette forholdet må jeg se etter en homolog karakter, for eksempel tilstedeværelsen av hår, brystkjertler og tre små bein i mellomøret - bare for å nevne noe. Imidlertid er homologier ikke enkle å diagnostisere.
Typer trær
Ikke alle trær er like, det er forskjellige grafiske fremstillinger, og hver og en klarer å innlemme noen særegne egenskaper ved gruppens evolusjon.
De mest grunnleggende trærne er kladogrammer. Disse grafene viser forholdet når det gjelder felles forfedre (i henhold til de siste vanlige forfedrene).
Tilsetnings trær inneholder tilleggsinformasjon og er representert i lengden på grenene.
Tallene assosiert med hver gren tilsvarer noen attributter i sekvensen - for eksempel mengden evolusjonær endring som organismer har gjennomgått. I tillegg til "additivtrær", er de også kjent som metriske trær eller fylogrammer..
Ultrametriske trær, også kalt dendogrammer, er et spesielt tilfelle av additivtrær, hvor tuppen av treet er like langt fra roten til treet.
Disse to siste variantene har alle dataene vi kan finne i et kladogram, og ekstra informasjon. Derfor er de ikke eksklusive, om ikke komplementære.
Politomias
Mange ganger er ikke noden på trærne fullstendig løst. Visuelt sies det at det er en polytomi når mer enn tre grener dukker opp fra en ny (det er en forfader for mer enn to nærmeste etterkommere). Når et tre ikke har polytomier, sies det å være fullstendig løst.
Det er to typer polytomier. Den første er de "harde" polytomiene. Disse er iboende for studiegruppen, og indikerer at etterkommerne utviklet seg samtidig. Alternativt indikerer "myke" polytomier uløste forhold forårsaket av data per se.
Evolusjonær klassifisering
Monofyletiske linjer
Evolusjonsbiologer søker å finne en klassifisering som passer til forgreningsmønsteret til fylogenetisk historie til grupper. I denne prosessen er det utviklet en rekke begreper som er mye brukt i evolusjonær biologi: monofyletisk, parafyletisk og polyfyletisk..
En monofyletisk taxon eller avstamning er en som består av en forfedres art, som er representert i noden, og alle dens etterkommere, men ikke andre arter. Denne grupperingen kalles en klade.
Monofyletiske linjer er definert på hvert nivå av taksonomisk hierarki. For eksempel anses Family Felidae, en avstamning som inneholder katter (inkludert huskatter), som monofyletisk..
Tilsvarende er Animalia også et monofyletisk takson. Som vi ser familien Felidae er innenfor Animalia, kan monofyletiske grupper hekkes.
Parafyletiske og polyfyletiske linjer
Imidlertid deler ikke alle biologer kladistisk klassifiseringstenkning. I tilfeller der dataene ikke er komplette eller bare for enkelhets skyld, blir visse taxa navngitt som inkluderer arter fra forskjellige klader eller høyere taxa som ikke deler en nyere felles forfader..
På denne måten er en taxon polyfyletisk, den defineres som en gruppe som inkluderer organismer fra forskjellige klader, og disse deler ikke en felles forfader. For eksempel, hvis vi ønsker å utpeke en gruppe homeotherms, vil det omfatte fugler og pattedyr..
I kontrast inneholder ikke en parafyletisk gruppe alle etterkommerne til den siste felles forfedren. Det ekskluderer med andre ord noen av medlemmene i gruppen. Det mest brukte eksemplet er reptiler, denne gruppen inneholder ikke alle etterkommerne til den siste felles forfedren: fugler.
applikasjoner
I tillegg til å bidra til den tøffe oppgaven med å belyse livets tre, har fylogenier også noen ganske viktige bruksområder.
På det medisinske feltet brukes fylogenier for å spore opprinnelse og smittehastigheter for smittsomme sykdommer, som AIDS, denguefeber og influensa..
De brukes også innen bevaringsbiologi. Kunnskap om fylogenien til en truet art er viktig for å spore kryssingsmønstre og nivået på hybridisering og innavl mellom individer..
Referanser
- Baum, D. A., Smith, S. D., og Donovan, S. S. (2005). Treetenkingen utfordring. Vitenskap, 310(5750), 979-980.
- Curtis, H., & Barnes, N. S. (1994). Invitasjon til biologi. Macmillan.
- Hall, B. K. (red.). (2012). Homologi: Det hierarkiske grunnlaget for komparativ biologi. Akademisk presse.
- Hickman, C. P., Roberts, L. S., Larson, A., Ober, W. C., og Garrison, C. (2001). Integrerte prinsipper for zoologi. McGraw-Hill.
- Hinchliff, CE, Smith, SA, Allman, JF, Burleigh, JG, Chaudhary, R., Coghill, LM, Crandall, KA, Deng, J., Drew, BT, Gazis, R., Gude, K., Hibbett, DS, Katz, LA, Laughinghouse, HD, McTavish, EJ, Midford, PE, Owen, CL, Ree, RH, Rees, JA, Soltis, DE, Williams, T.,… Cranston, KA (2015). Syntese av fylogeni og taksonomi til et omfattende livets tre. Proceedings of the National Academy of Sciences i De forente stater, 112(41), 12764-9.
- Kardong, K. V. (2006). Vertebrater: komparativ anatomi, funksjon, evolusjon. McGraw-Hill.
- Page, R. D., & Holmes, E. C. (2009). Molekylær evolusjon: en fylogenetisk tilnærming. John Wiley & Sons.



Ingen har kommentert denne artikkelen ennå.